Détection des noyaux et quantification de fluorescence dans Python: un guide étape par étape (partie 2)



Auteur (s): MicrobioscopicData (par Alexandros Athanasopoulos)
Publié à l’origine sur Vers l’IA.
Bienvenue dans le deuxième tutoriel de notre série, «Détection des noyaux et quantification de fluorescence dans Python». Dans ce tutoriel, nous nous concentrerons sur la mesure de l’intensité de fluorescence du canal GFP, l’extraction de données pertinentes et la réalisation d’une analyse détaillée pour dériver des informations biologiques significatives.
Pour bénéficier pleinement de ce tutoriel, il est utile d’avoir une compréhension de base de la programmation Python ainsi que de la familiarité avec la microscopie à fluorescence, y compris les principes derrière l’utilisation de protéines fluorescentes comme la GFP (protéine fluorescente verte).
Dans le tutoriel précédent, nous avons utilisé des images de cellules de fibroblastes où les noyaux sont marqués avec du DAPI, un colorant fluorescent (canal bleu) qui se lie à l’ADN et une protéine d’intérêt présente à la fois dans le cytoplasme et le noyau, détecté dans le canal vert. Nous avons commencé par prétraiter les images pour améliorer la qualité des données. Nous avons appliqué le lissage gaussien avec des valeurs sigma variables pour réduire le bruit et utilisé des méthodes de seuillage pour distinguer efficacement les noyaux de l’arrière-plan. De plus, nous avons discuté des techniques de post-traitement, telles que la suppression de petits artefacts, pour affiner davantage les résultats de la segmentation.
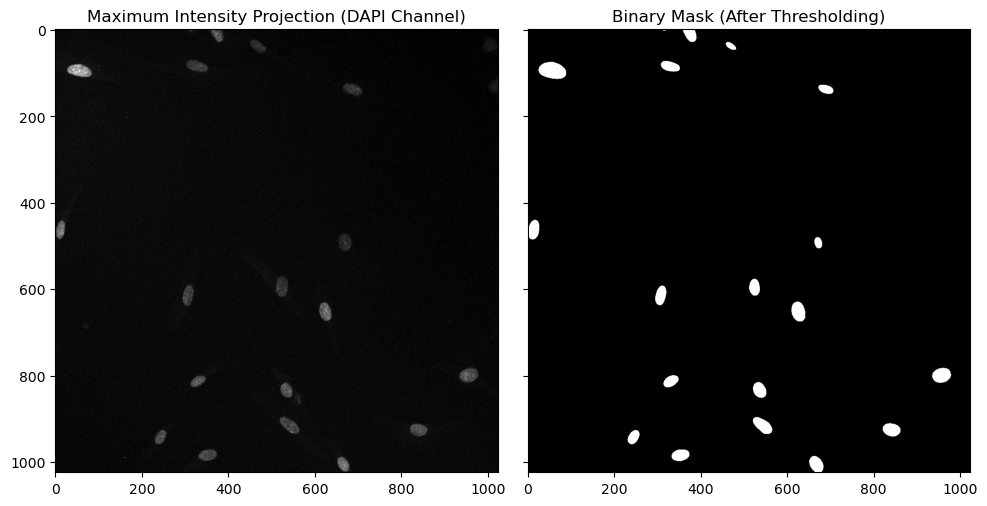
Le code ci-dessous (à partir de notre premier tutoriel) segmente et visualise efficacement les noyaux dans les images de microscopie à fluorescence, offrant des informations claires sur la distribution et l’intensité des caractéristiques détectées. L’étape suivante de la quantification de la fluorescence consiste à étiqueter les noyaux segmentés.
from skimage import io, filters, morphology, measure, segmentation, color
from skimage.measure import regionprops
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
import seaborn as sns
# Set option to display all columns and rows in Pandas DataFrames
pd.set_option('display.max_columns', None)
pd.set_option('display.max_rows', None)# Load the multi-channel TIFF image
image = io.imread('fibro_nuclei.tif')
# Separate the GFP channel (assuming channel 0 is GFP)
channel1 = image(:, 0, :, :) # GFP channel
# Perform Maximum Intensity Projection (MIP) on GFP channel
channel1_max_projection = np.max(channel1, axis=0)
# Separate the DAPI channel (assuming channel 1 is DAPI)
channel2 = image(:, 1, :, :) # DAPI channel
# Perform Maximum Intensity Projection (MIP) on DAPI channel
channel2_max_projection = np.max(channel2, axis=0)
# Apply Gaussian smoothing to the DAPI MIP
smoothed_image = filters.gaussian(channel2_max_projection, sigma=5)
# Apply Otsu's method to find the optimal threshold and create a binary mask
threshold_value = filters.threshold_otsu(smoothed_image)
binary_mask = smoothed_image > threshold_value
# Create subplots with shared x-axis and y-axis
fig, (ax1, ax2) = plt.subplots(1, 2, sharex=True, sharey=True, figsize=(10, 10))
# Visualize the Maximum Intensity Projection (MIP) for the DAPI channel
ax1.imshow(channel2_max_projection, cmap='gray')
ax1.set_title('Maximum Intensity Projection (DAPI Channel)')
# Visualize the binary mask obtained after thresholding the smoothed DAPI MIP
ax2.imshow(binary_mask, cmap='gray')
ax2.set_title('Binary Mask (After Thresholding)')
# Adjust layout to prevent overlap
plt.tight_layout()
# Display the plots
plt.show()
Étiquetage des noyaux segmentés
L’étiquetage du masque binaire est une étape cruciale dans l’analyse d’image. Lorsque nous effectuons un seuil sur une image, le résultat est un masque binaire (voir également notre tutoriel précédent) où les pixels sont classés comme premier plan / vrai (par exemple, noyaux) ou arrière-plan / faux. Cependant, ce masque binaire seul ne fait pas de distinction entre différents noyaux individuels – il montre simplement quels pixels appartiennent au premier plan et à l’arrière-plan.
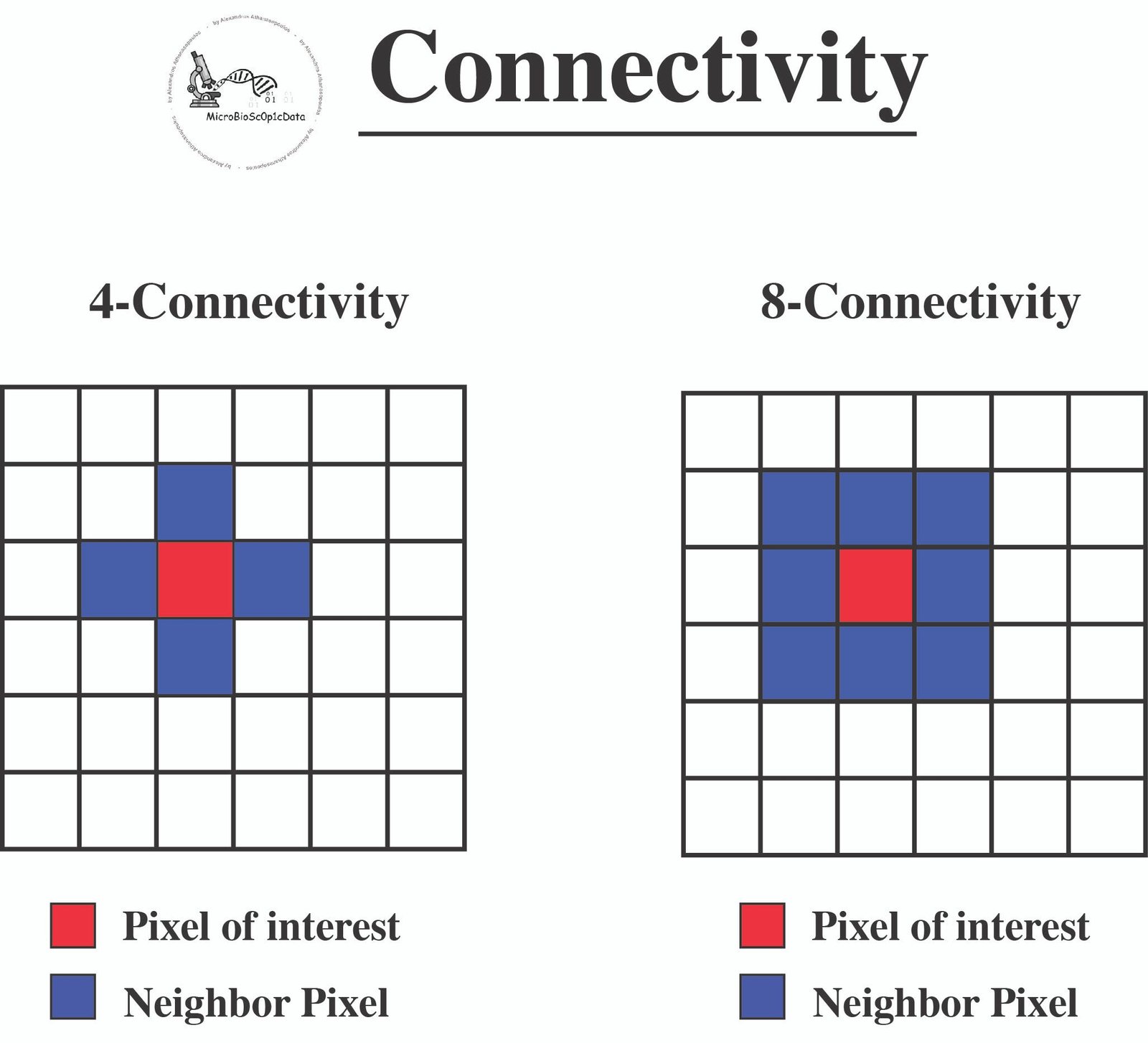
Étiquetage est le processus d’attribution d’un identifiant unique (étiquette) à chaque noyau du masque binaire. Dans le contexte des composants connectés, l’étiquetage implique Identification et marquage des groupes de pixels connectés (composants) Cela représente des objets individuels, comme les noyaux, dans l’image. Une fois le masque binaire créé, l’algorithme des composants connectés est appliqué. Cet algorithme scanne le masque binaire pour détecter des groupes de pixels connectés en utilisant des critères de connectivité à 4 ou 8-connectivité (voir ci-dessous l’image) et attribue une étiquette unique à chaque composant connecté. Chaque étiquette correspond à un noyau distinct de l’image (1).
Il existe différents types de connectivité, principalement la 4-connectivité et la 8-connectivité:
4-connectivité:
- Définition: En 4-Connectivité, un pixel (d’intérêt) est considéré comme connecté à un autre pixel s’ils partagent un bord. Dans une grille 2D, chaque pixel a quatre voisins possibles: à gauche, à droite, au-dessus et en dessous.
- Applications: La connectivité 4 est souvent utilisée dans les algorithmes où les connexions diagonales ne sont pas considéréesfournissant ainsi une forme de connectivité plus restrictive.
8-connectivité:
- Définition: En 8-connectivité, un pixel (d’intérêt) est lié à tous ses voisins, y compris ceux qui partagent un sommet. Cela signifie que, en plus des quatre voisins connectés à bord (comme dans la 4-connectivité), le pixel est également connecté aux quatre voisins diagonaux.
- Applications: La connectivité 8 est utilisée dans les applications où les connexions diagonales sont significatives, fournissant une forme de connectivité plus inclusive.
Pourquoi l’étiquetage est important
- Identification: L’étiquetage nous permet d’identifier et de différencier les noyaux individuels dans le masque binaire. Chaque noyau a une étiquette unique, ce qui permet de traiter et d’analyser chaque noyau séparément.
- Analyse: Une fois que les noyaux sont marqués, nous pouvons mesurer diverses propriétés de chaque noyau individuellement, comme la surface, le périmètre et l’intensité de la fluorescence… Ceci est essentiel pour l’analyse quantitative dans la recherche biologique.
- Visualisation: L’étiquetage facilite également la visualisation des noyaux segmentés. En attribuant différentes couleurs ou intensités à chaque étiquette, nous pouvons facilement voir et distinguer les noyaux segmentés dans une image étiquetée.
Le code ci-dessous est utilisé pour étiqueter les régions connectées (composants) dans notre image binaire. La fonction skimage.measure.label scanne le masque binaire et attribue une étiquette entière unique à chaque composant connecté. La sortie est une image étiquetée (tableau 2D Numpy) où chaque composant connecté se voit attribuer une étiquette entier unique (par exemple, 1, 2, 3, etc.). Les pixels qui appartiennent au même composant (par exemple, un seul noyau) auront la même étiquette. Par défaut, la fonction utilise la 8-connectivité.
La fonction color.label2rgb(labeled_nuclei, bg_label=0) du module SKIMage.color convertit une image étiquetée en une image RVB (couleur).
labeled_nuclei: Ceci est l’image étiquetéebg_label=0: Cela spécifie que l’étiquette de fond est 0, donc l’arrière-plan ne sera pas coloré, et seules les régions marquées (noyaux) seront colorées différemment dans l’image RVB de sortie.
Le segmentation.clear_border() La fonction est utilisée ensuite pour éliminer tous les noyaux qui touchent les bords de l’image, garantissant que Seuls les noyaux entièrement contenus sont considérés. L’image est ensuite réétiquetée pour refléter l’élimination de ces noyaux de touche à la bordure, et le nombre mis à jour est imprimé. Enfin, les noyaux marqués sont visualisés en couleur, avec chaque noyau annoté à son centroïde en utilisant son numéro d’étiquette correspondant.
# Label the nuclei and return the number of labeled components
labeled_nuclei, num_nuclei = measure.label(binary_mask, return_num=True)print(f"Initial number of labeled nuclei: {num_nuclei}")
# Remove nuclei that touch the borders
cleared_labels = segmentation.clear_border(labeled_nuclei)# Recalculate the number of labeled nuclei after clearing the borders
# Note: We need to exclude the background (label 0)
final_labels, final_num_nuclei = measure.label(cleared_labels > 0, return_num=True)print(f"Number of labeled nuclei after clearing borders: {final_num_nuclei}")
# Visualize the labeled nuclei
plt.figure(figsize=(10, 10))
plt.imshow(color.label2rgb(final_labels, bg_label=0), cmap='nipy_spectral')
plt.title('Labeled Nuclei')
plt.axis('off')# Annotate each nucleus with its label
for region in measure.regionprops(final_labels):
# Take the centroid of the region and use it for placing the label
y, x = region.centroid
plt.text(x, y+30, f"Nucleus: {region.label}", color='white', fontsize=12, ha='center', va='center')plt.show()
Initial number of labeled nuclei: 19
Number of labeled nuclei after clearing borders: 15
Mesurer la fluorescence
Pour mesurer la fluorescence dans le canal vert (GFP) de notre image de pile Z multi-canaux, nous additionnons les valeurs de pixels du canal GFP dans les régions définies par notre masque binaire, au lieu de s’appuyer uniquement sur la projection d’intensité maximale.
Cette méthode (en somme des valeurs de pixels) fournit une meilleure représentation du signal de fluorescence total dans chaque région marquée (noyau) car elle explique toute la distribution d’intensité plutôt que simplement les pixels les plus brillants.
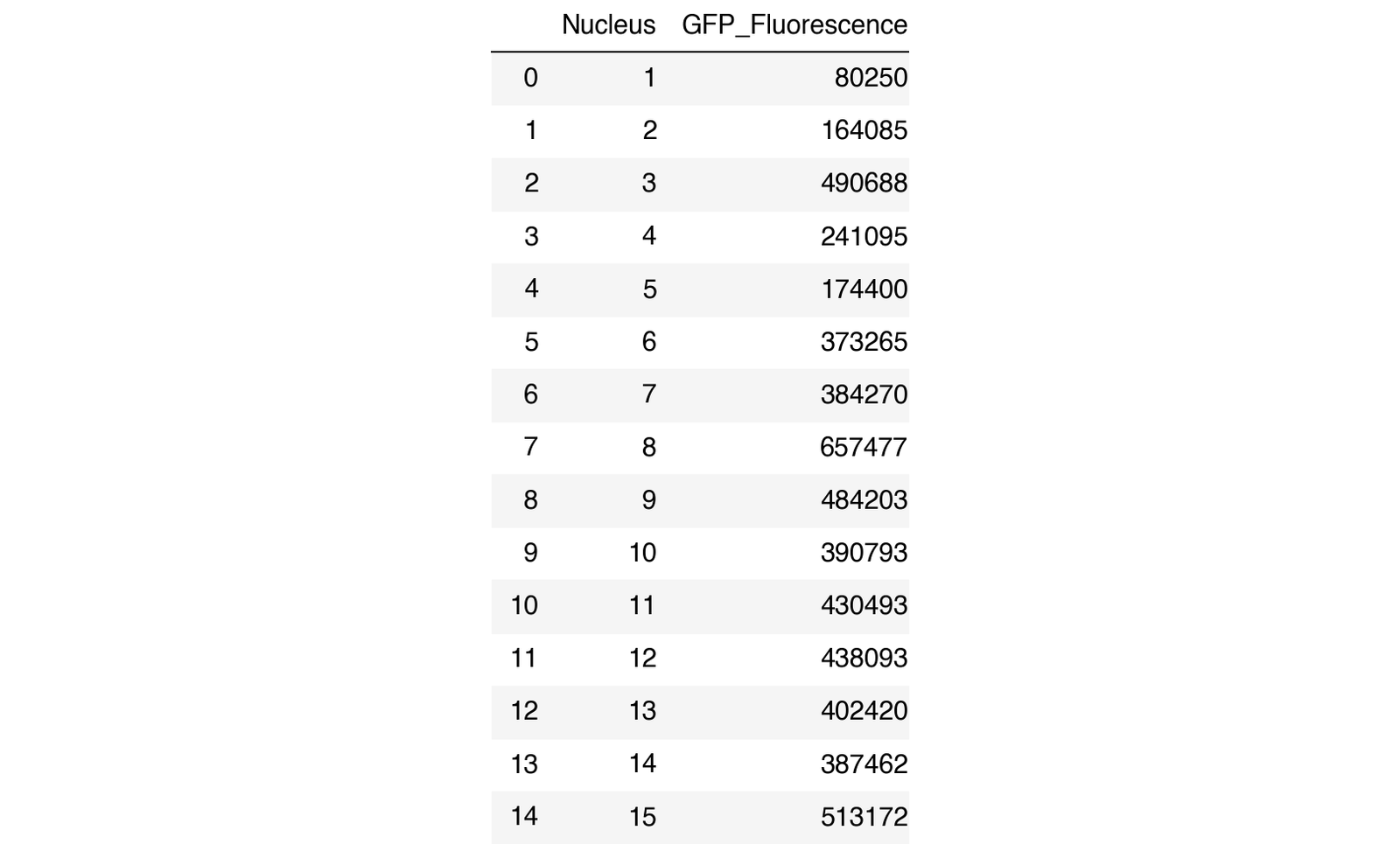
Le code ci-dessous calcule la fluorescence GFP totale pour chaque noyau marqué dans l’image en additionnant les intensités de pixels dans le canal GFP. Les valeurs résultantes sont stockées dans une liste pour une analyse plus approfondie, comme la comparaison de la fluorescence à travers différents noyaux ou l’évaluation de la distribution de GFP dans l’échantillon. L’opération channel1.sum(axis=0) résume les intensités de pixels sur toutes les zones Z pour chaque position (x, y) dans l’image. Il en résulte une image 2D où chaque valeur de pixel représente l’intensité de fluorescence totale à cette coordonnée (x, y) à travers la profondeur entière de l’échantillon.
# Sum fluorescence in GFP channel within each labeled nucleus
gfp_fluorescence = ()for region in measure.regionprops(final_labels, intensity_image=channel1.sum(axis=0)): # channel1.sum(axis=0) has a data type of 64-bit unsigned integer
gfp_sum = region.intensity_image.sum()
gfp_fluorescence.append(gfp_sum)# Print the total fluorescence for each nucleus
Nucleus 1: Total GFP Fluorescence = 80250
for i, fluorescence in enumerate(gfp_fluorescence, start=1):
print(f"Nucleus {i}: Total GFP Fluorescence = {fluorescence}")
Nucleus 2: Total GFP Fluorescence = 164085
Nucleus 3: Total GFP Fluorescence = 490688
Nucleus 4: Total GFP Fluorescence = 241095
Nucleus 5: Total GFP Fluorescence = 174400
Nucleus 6: Total GFP Fluorescence = 373265
Nucleus 7: Total GFP Fluorescence = 384270
Nucleus 8: Total GFP Fluorescence = 657477
Nucleus 9: Total GFP Fluorescence = 484203
Nucleus 10: Total GFP Fluorescence = 390793
Nucleus 11: Total GFP Fluorescence = 430493
Nucleus 12: Total GFP Fluorescence = 438093
Nucleus 13: Total GFP Fluorescence = 402420
Nucleus 14: Total GFP Fluorescence = 387462
Nucleus 15: Total GFP Fluorescence = 513172
Analyse des données
Le code ci-dessus a pratiquement calculé la densité intégrée qui est une mesure utilisée dans l’analyse d’image pour quantifier la quantité de signal (par exemple, fluorescence) dans une région d’intérêt (comme un noyau).
En microscopie à fluorescence, la densité intégrée peut être utilisée pour estimer la quantité totale de fluorescence dans un noyau ou un compartiment cellulaire donné. Cela peut être utile pour comparer les niveaux d’expression d’une protéine marquée par fluorescence entre différentes cellules ou des conditions expérimentales.
Le code ci-dessous convertit le gfp_fluorescence list en un pandas dataframe Pour une analyse statistique supplémentaire, comme la comparaison de la fluorescence à travers différents noyaux ou conditions, calculer la moyenne et l’écart type, ou effectuer des analyses plus avancées comme le clustering ou corrélation études.
# Convert the fluorescence data into a DataFrame
df = pd.DataFrame({'Nucleus': range(1, len(gfp_fluorescence) + 1), 'GFP_Fluorescence': gfp_fluorescence})# Display the DataFrame
df
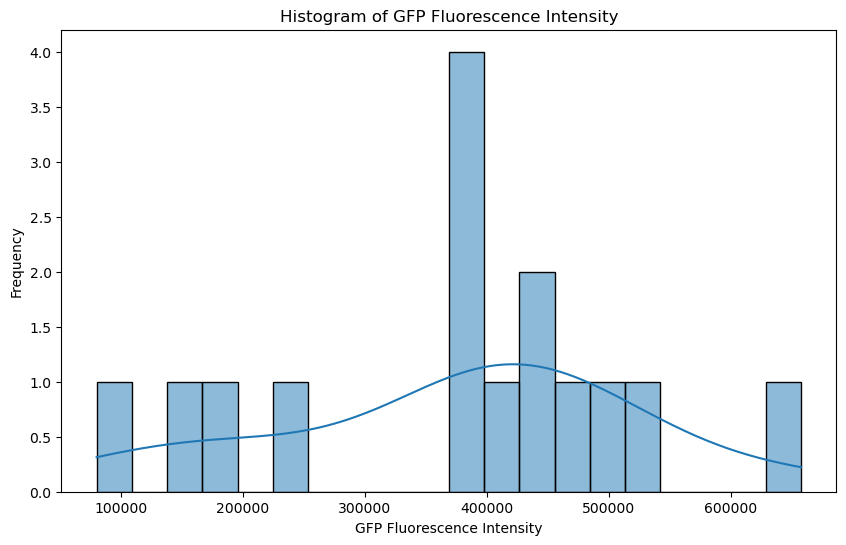
En analysant la distribution de l’intensité de fluorescence à travers les noyaux, nous pouvons potentiellement révéler la présence de différentes populations ou sous-groupes dans l’échantillon. Cette analyse pourrait fournir des informations précieuses, telles que l’identification des modèles d’expression ou des réponses distinctes au traitement. Des techniques comme le clustering peuvent aider à catégoriser les noyaux en fonction de leurs profils de fluorescence, permettant des interprétations biologiques plus profondes.
# Plot histogram
plt.figure(figsize=(10, 6))
sns.histplot(df('GFP_Fluorescence'), bins=20, kde=True)
plt.title('Histogram of GFP Fluorescence Intensity')
plt.xlabel('GFP Fluorescence Intensity')
plt.ylabel('Frequency')
plt.show()
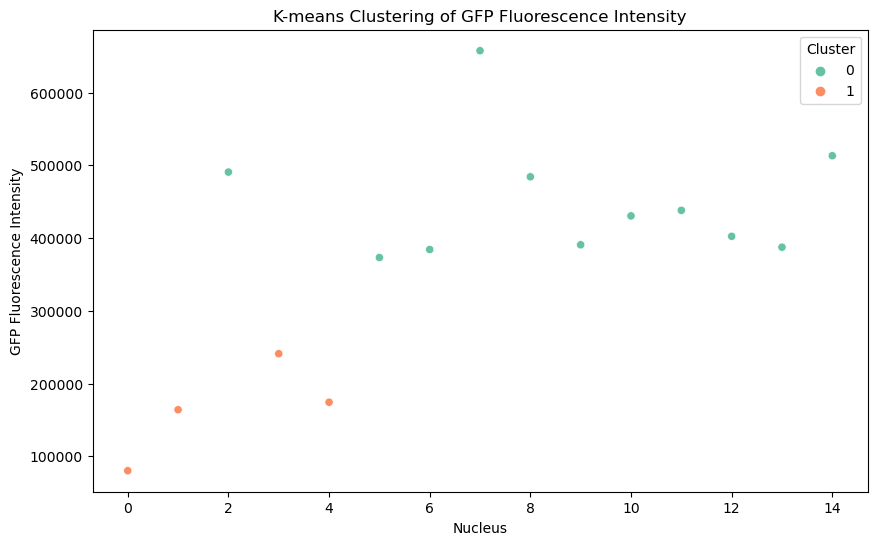
Analyse de clustering:
Nous pouvons appliquer le clustering K-Means pour regrouper les noyaux en fonction de leur intensité de fluorescence. Cela peut aider à identifier des populations distinctes qui diffèrent dans leurs niveaux d’expression. Dans le tracé de diffusion en dessous de chaque point représente un noyau, avec l’axe des x montrant l’indice du noyau et l’axe y montrant l’intensité totale de fluorescence GFP pour ce noyau. Les points sont codés en couleur en fonction du cluster auquel ils appartiennent. Deux clusters sont représentés: le cluster 0 (en vert) et le cluster 1 (en orange). Le regroupement a été effectué en utilisant des k-means avec deux clusters. Ce tracé montre comment les noyaux peuvent être regroupés en grappes distinctes en fonction de leur intensité de fluorescence GFP.
from sklearn.cluster import KMeans# Reshape data for clustering
fluorescence_data = df('GFP_Fluorescence').values.reshape(-1, 1)
# Apply K-means clustering (let's assume 2 clusters for simplicity)
kmeans = KMeans(n_clusters=2, random_state=0).fit(fluorescence_data)
df('Cluster') = kmeans.labels_
# Visualize clusters
plt.figure(figsize=(10, 6))
sns.scatterplot(x=df.index, y=df('GFP_Fluorescence'), hue=df('Cluster'), palette='Set2')
plt.title('K-means Clustering of GFP Fluorescence Intensity')
plt.xlabel('Nucleus')
plt.ylabel('GFP Fluorescence Intensity')
plt.show()
Ensemble, ces graphiques (histogramme et tracé de diffusion) indiquent la présence d’au moins deux sous-populations de noyaux en fonction de leur fluorescence GFP, reflétant potentiellement la variabilité biologique ou des conditions différentes affectant l’expression de fluorescence.
Conclusion
Dans ce tutoriel, nous avons exploré des techniques de traitement d’image avancées pour segmenter les noyaux et quantifier les signaux fluorescents à l’aide de Python. En utilisant des méthodes comme le lissage gaussien, le seuil et le marquage des composants connectés, nous avons pu identifier avec précision et séparer les noyaux individuels dans le canal DAPI. Nous avons également démontré comment mesurer l’intensité de la fluorescence dans le canal GFP en additionnant les valeurs de pixels à travers les z-Slices pour capturer la distribution complète de la fluorescence dans chaque noyau. Grâce à l’analyse des données, nous avons pu quantifier et interpréter les signaux de fluorescence, permettant des informations plus approfondies sur les variations biologiques.
Références:
(1) P. Bankhead, «Introduction à l’analyse des bioimages – Introduction à l’analyse du bioimage». https://bioimagebook.github.io/index.html (consulté le 29 juin 2023).
Publié via Vers l’IA
Source link