Outils d’intégration spécifiques à la santé pour la dermatologie et la pathologie






Il y a une pénurie mondiale d’accès à l’interprétation des experts en imagerie médicale à travers les spécialités, notamment radiologie, dermatologie et pathologie. La technologie d’apprentissage automatique (ML) peut aider à soulager ce fardeau en alimentant les outils qui permettent aux médecins d’interpréter ces images plus précisément et efficacement. Cependant, le développement et la mise en œuvre de ces outils ML sont souvent limités par la disponibilité de données de haute qualité, d’expertise en ML et de ressources informatiques.
Une façon de catalyser l’utilisation de la ML pour l’imagerie médicale est via des modèles spécifiques au domaine qui utilisent l’apprentissage en profondeur (DL) pour capturer les informations dans les images médicales en tant que vecteurs numériques compressés (appelés intégres). Ces intérêts représentent un type de compréhension pré-appris des caractéristiques importantes d’une image. L’identification des modèles dans les intérêts réduit la quantité de données, d’expertise et de calcul nécessaire pour former des modèles performants par rapport à Travailler avec des données de grande dimensioncomme les images, directement. En effet, ces intérêts peuvent être utilisés pour effectuer une variété de tâches en aval dans le domaine spécialisé (voir graphique animé ci-dessous). Ce cadre de mise à profit de la compréhension pré-appris pour résoudre les tâches connexes est similaire à celle d’un guitariste chevronné apprenant rapidement une nouvelle chanson par Ear. Parce que le guitariste a déjà construit une base de compétences et de compréhension, il peut rapidement prendre les motifs et le groove d’une nouvelle chanson.
Afin de rendre ce type de modèle d’incorporation et de stimuler le développement des outils ML dans l’imagerie médicale, nous sommes ravis de publier deux outils spécifiques au domaine pour l’utilisation de la recherche: Fondation derm et Fondation Path. Cela suit la forte réponse que nous avons déjà reçue des chercheurs en utilisant le Fondation CXR L’outil d’incorporation pour les radiographies thoraciques et représente une partie de nos offres de recherche en expansion sur plusieurs modalités spécialisées médicales. Ces outils d’incorporation prennent une image en entrée et produisent respectivement un vecteur numérique (l’incorporation) spécialisé dans les domaines des images de dermatologie et de pathologie numérique. En exécutant un ensemble de données de radiographie thoracique, de dermatologie ou d’images de pathologie à travers l’outil d’incorporation respectif, les chercheurs peuvent obtenir des intégres pour leurs propres images et utiliser ces intérêts pour développer rapidement de nouveaux modèles pour leurs applications.
Fondation Path
Dans « Optimisation spécifique au domaine et évaluation diversifiée des modèles auto-supervisés pour l’histopathologie», Nous avons montré que les modèles d’apprentissage auto-supervisé (SSL) pour les images de pathologie surpassent les approches pré-formation traditionnelles et permettent une formation efficace des classificateurs pour les tâches en aval. Cet effort s’est concentré sur hématoxyline et éosine (H&E) Diapositives colorées, la coloration des tissus principaux en pathologie diagnostique qui permet aux pathologistes de visualiser les caractéristiques cellulaires au microscope. Les performances des classificateurs linéaires formées à l’aide de la sortie des modèles SSL correspondaient à celles des modèles DL antérieurs formés aux ordres de grandeur des données plus étiquetées.
En raison de différences substantielles entre les images de pathologie numérique et les photos de «l’image naturelle», ce travail a impliqué plusieurs optimisations spécifiques à la pathologie pendant la formation des modèles. Un élément clé est que Images à glissement entier (WSIS) en pathologie peut être de 100 000 pixels de diamètre (des milliers de fois plus grand que les photos de smartphone typiques) et sont analysés par des experts à plusieurs grossissements (niveaux de zoom). En tant que tels, les WSIS sont généralement décomposés en carreaux ou correctifs plus petits pour la vision par ordinateur et les applications DL. Les images résultantes sont des informations denses avec des cellules ou des structures tissulaires distribuées dans tout le cadre au lieu d’avoir des objets sémantiques distincts ou des variations de premier plan par rapport aux fondations, créant ainsi des défis uniques pour une SSL robuste et une extraction de caractéristiques. De plus, physique (par exemple, coupe) et chimique (par exemple, fixation et tasage) Les processus utilisés pour préparer les échantillons peuvent influencer considérablement l’apparence de l’image.
En tenant compte de ces aspects importants, les optimisations SSL spécifiques à la pathologie ont inclus d’aider le modèle à apprendre Caractéristiques de la tachegénéralisation du modèle à des correctifs à partir de multiples grossissements, augmentation Les données pour imiter la numérisation et le post-traitement d’image et l’équilibrage des données personnalisés pour améliorer l’hétérogénéité des entrées pour la formation SSL. Ces approches ont été largement évaluées à l’aide d’un large ensemble de tâches de référence impliquant 17 types de tissus différents sur 12 tâches différentes.
En utilisant le transformateur de vision (Vit-S / 16) Architecture, Path Foundation a été sélectionné comme modèle le plus performant du processus d’optimisation et d’évaluation décrit ci-dessus (et illustré dans la figure ci-dessous). Ce modèle fournit donc un équilibre important entre les performances et la taille du modèle pour permettre une utilisation précieuse et évolutive pour générer des intérêts sur les nombreux patchs d’image individuels de grande pathologie WSIS.
 |
| Formation SSL avec des optimisations spécifiques à la pathologie pour la Fondation Path. |
La valeur des représentations d’images spécifiques au domaine peut également être observée dans la figure ci-dessous, qui montre l’amélioration des performances de sondage linéaire de la fondation du chemin (tel que mesuré par Auc) par rapport à la pré-formation traditionnelle sur les images naturelles (Imagenet-21k). Cela comprend l’évaluation des tâches telles que Détection métastatique du cancer du sein dans les ganglions lymphatiques, classement du cancer de la prostateet classement du cancer du seinentre autres.
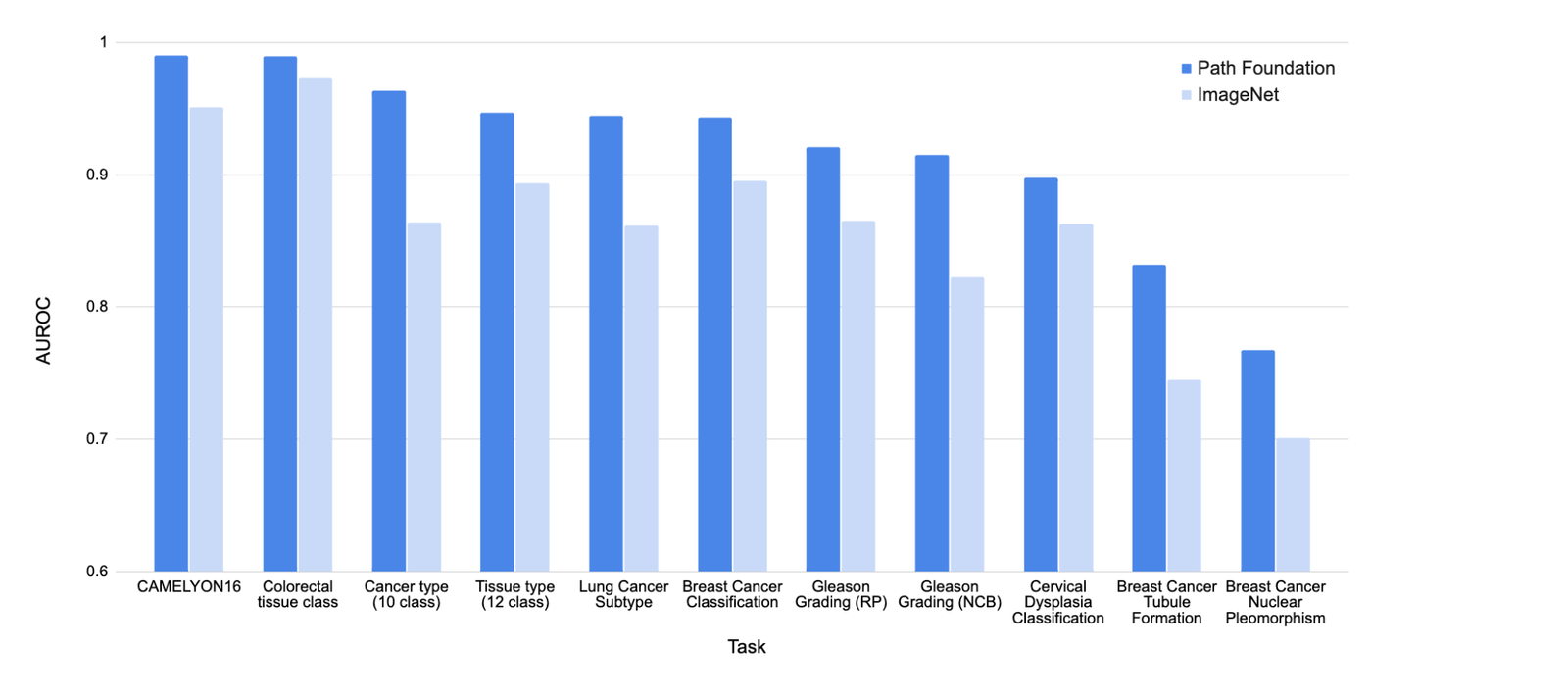 |
| Les intérêts des fondations de PATH surpassent considérablement les intégres d’imaget traditionnels, comme évalué par sondage linéaire sur plusieurs tâches d’évaluation en histopathologie. |
Fondation derm
Fondation derm est un outil d’intégration dérivé de nos recherches sur l’application de DL Interpréter les images des conditions de dermatologie et comprend nos travaux récents qui ajoutent Améliorations pour mieux généraliser aux nouveaux ensembles de données. En raison de sa pré-formation spécifique à la dermatologie, il a une compréhension latente des caractéristiques présentes dans les images des conditions cutanées et peut être utilisée pour développer rapidement des modèles pour classer les affections cutanées. Le modèle sous-jacent à l’API est un Bit resnet-101×3 formé en deux étapes. La première étape de pré-formation utilise l’apprentissage contrasté, similaire à Convavepour s’entraîner sur un grand nombre de paires de texte d’image depuis Internet. Dans la deuxième étape, la composante d’image de ce modèle pré-formé est ensuite affinée pour la classification des conditions à l’aide d’ensembles de données cliniques, tels que ceux des services de télédermatologie.
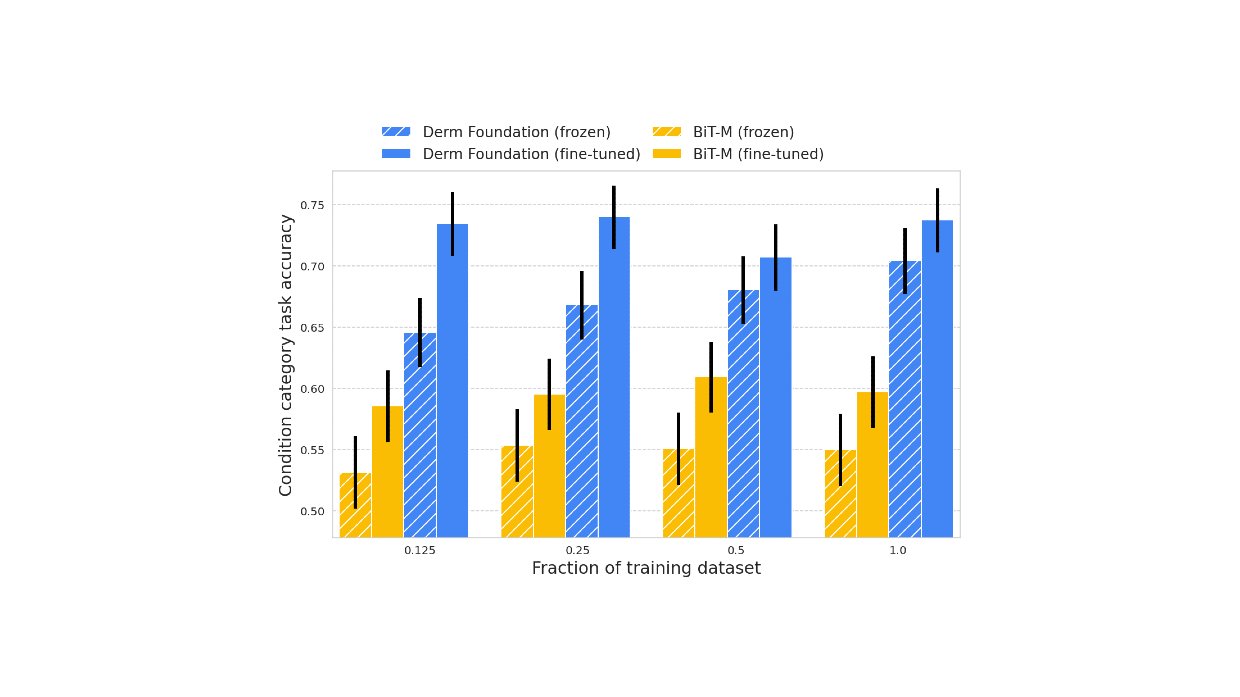
Contrairement aux images histopathologiques, les images de dermatologie ressemblent plus étroitement aux images du monde réel utilisées pour former de nombreux modèles de vision informatique d’aujourd’hui. Cependant, pour les tâches de dermatologie spécialisées, la création d’un modèle de haute qualité peut encore nécessiter un grand ensemble de données. Avec Derm Foundation, les chercheurs peuvent utiliser leur propre ensemble de données plus petit pour récupérer des intégres spécifiques au domaine et les utiliser pour construire des modèles plus petits (par exemple, des classificateurs linéaires ou d’autres petits modèles non linéaires) qui leur permettent de valider leurs idées de recherche ou de produit. Pour évaluer cette approche, nous avons formé des modèles sur une tâche en aval à l’aide de données de télédermatologie. La formation du modèle impliquait des tailles d’ensemble de données variables (12,5%, 25%, 50%, 100%) pour comparer les classificateurs linéaires basés sur l’intégration contre le réglage fin.
Les variantes de modélisation considérées étaient:
- Un classificateur linéaire sur des intégres intégrés de Bit-m (un modèle d’image pré-formé standard)
- Version affinée de Bit-M avec une couche supplémentaire dense pour la tâche en aval
- Un classificateur linéaire sur les incorporations gelées de l’API Derm Foundation
- Version affinée du modèle sous-jacente à l’API Derm Foundation avec une couche supplémentaire pour la tâche en aval
Nous avons constaté que des modèles construits sur les intérimaires Derm Foundation pour les tâches liés à la dermatologie ont obtenu une qualité significativement plus élevée que celles construites uniquement sur les intérêts ou ajustés fins à partir de Bit-M. Cet avantage s’est avéré le plus prononcé pour les tailles de jeu de données de formation plus petites.
Cependant, il y a des limites à cette analyse. Nous explorons toujours dans quelle mesure ces intérêts se généralisent entre les types de tâches, les populations de patients et les paramètres d’image. Les modèles en aval construits à l’aide de Derm Foundation nécessitent toujours une évaluation minutieuse pour comprendre leurs performances attendues dans le cadre prévu.
Chemin d’accès et fondation Derm
Nous envisageons que les outils d’intégration de la fondation Derm et de la Fondation Path permettront une gamme de cas d’utilisation, notamment le développement efficace de modèles de tâches de diagnostic, l’assurance qualité et les améliorations pré-analytiques du flux de travail, l’indexation et la conservation d’image et la découverte et la validation des biomarqueurs. Nous publions les deux outils à la communauté de la recherche afin qu’ils puissent explorer l’utilité des intérêts pour leurs propres données de dermatologie et de pathologie.
Pour accéder, veuillez vous inscrire aux conditions d’utilisation de chaque outil à l’aide des formulaires Google suivants.
Après avoir accédé à chaque outil, vous pouvez utiliser l’API pour récupérer des intégres à partir d’images de dermatologie ou d’images de pathologie numérique stockées dans Google Cloud. Les utilisateurs approuvés qui sont juste curieux de voir le modèle et les intégres en action peuvent utiliser l’exemple fourni des cahiers Colab pour former des modèles en utilisant des données publiques pour classer Six affections cutanées communes ou identifier les tumeurs dans patchs histopathologiques. Nous sommes impatients de voir la gamme des cas d’utilisation que ces outils peuvent débloquer.
Remerciements
Nous tenons à remercier les nombreux collaborateurs qui ont aidé à rendre ce travail possible, notamment Yun Liu, Can Kirmizi, Fereshteh Mahvar, Bram Sterling, Arman Tajback, Kenneth Philbrik, Arnav Agharwal, Aurora Cheung Ayush Jain, Amit Talreja, Rajeev Rikhye, Abbi Ward, Jeremy Lai, Faruk Ahmed, Supriya Vijay, Tiam Jaroensri, Jessica Loo, Saurabh Vyawahare, Saloni Agarwal, Ellery Wulczy Winer, Sami Lachgar, Yossi Matias, Greg Corrado et Dale Webster.
Source link